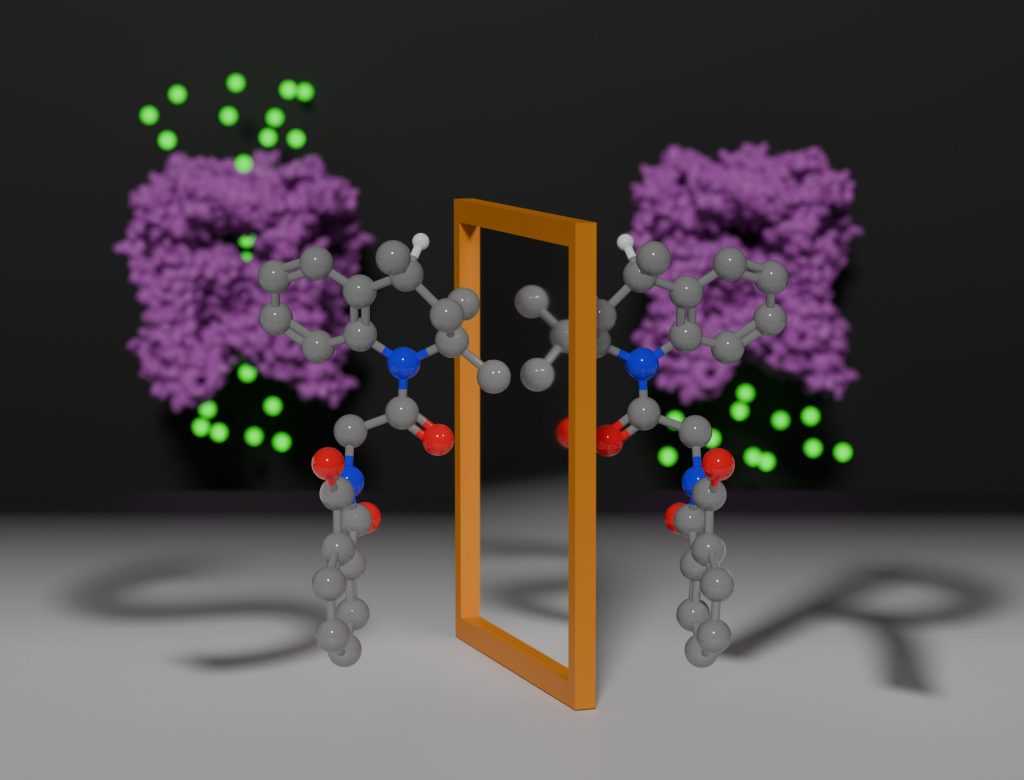
Uma etapa importante da pesquisa em química medicinal é encontrar moléculas que interajam diretamente com proteínas-alvo de forma potente e inequívoca, provocando uma cadeia de reações nas células. Este é um dos primeiros passos do processo de desenvolvimento de fármacos. Mas encontrar uma boa molécula não é suficiente. É de fundamental importância verificar se ela é um enantiômero, ou seja, se existem outras versões espelhadas da molécula, seja no plano horizontal, vertical ou em ambos.
O artigo foi publicado na revista ChemBioChem e é assinado por pesquisadores das universidades Estadual de Campinas (Unicamp), Federal de São Paulo (Unifesp), Federal de São Carlos (UFSCar), Federal Fluminense (UFF) e do Instituto Federal de Educação, Ciência e Tecnologia de São Paulo (IFSP).
Para melhor entendimento, vale recordar alguns conceitos básicos de biologia celular. A célula tem várias organelas, entre elas os lisossomos, responsáveis por “reciclar” os componentes celulares. Ao encontrar resíduos no citoplasma, a vesícula os envolve e os lança para seu interior, onde serão digeridos por meio de um processo chamado de autofagia. O mau funcionamento dessas proteínas pode causar doenças como mucolipidose, tumores e maior suscetibilidade a infecções.
Quando o objetivo da pesquisa é buscar um composto capaz de interagir com a proteína existente na membrana do lisossomo, a TRPML1, o sucesso vai depender de qual versão da molécula está interagindo com esse alvo. Como demonstram os autores do artigo, essa informação otimiza a busca do melhor composto para estudar a proteína-alvo envolvida na reciclagem celular e cujo mau funcionamento está relacionado a doenças.
Para ter uma ideia de como intervir nesse mecanismo celular pode ser relevante, em novembro de 2022 a empresa Casma Therapeutics, que trabalha na descoberta de novos fármacos, recebeu um investimento de US$ 46 milhões para o desenvolvimento de medicamentos focados na autofagia. Já o grupo Merck, que atua no setor de ciência e tecnologia, investiu mais de US$ 1 bilhão na compra de duas empresas dedicadas ao desenvolvimento de ativadores da proteína TRPML1.
Os autores já sabiam que a molécula ML-SA1 era capaz de interagir com a proteína de membrana para ativá-la. “Notamos que apenas parte das moléculas conseguia interagir e ativar com sucesso a proteína-alvo”, explica Micael Cunha, bolsista de pós-doutorado da FAPESP. A resposta veio após os pesquisadores estabelecerem um protocolo de separação dos enantiômeros S e R da ML-SA1 e constatarem que apenas um deles tem efeito na proteína, ainda que ambos compartilhem a mesma estrutura e o mesmo número de átomos.
Tal informação é de extrema importância para os estudos baseados em alvo, pois revela que parte das moléculas usadas para ativar a TRPML1 está na verdade competindo pelo espaço de encaixe na proteína com as moléculas que realmente a ativam. Quando a forma S é testada isoladamente, a ativação é dez vezes maior do que o composto em mistura, enquanto a forma R não apresenta atividade. Segundo os resultados obtidos pelos pesquisadores, o enantiômero (R)-ML-SA1 se liga no mesmo local que o S, porém sem efeito.
Antes de testar as moléculas do tipo S e R isoladamente, foi preciso separá-las. Bruno Amaral, pesquisador do Centro de Química Medicinal (CQMED) da Unicamp, trabalhou na separação dos enantiômeros utilizados na pesquisa e explica que foi necessário estabelecer um novo protocolo, testando diferentes temperaturas, colunas de cromatografia e solventes. A colaboração com a professora Quezia Cass, do Núcleo de Pesquisa em Cromatografia da UFSCar, possibilitou o desenvolvimento desse protocolo.
Depois de separadas, os pesquisadores definiram qual era a versão S e R. “Quando eu consegui uma boa separação, só sabíamos que eram formas diferentes, mas não era possível identificar qual era a S e qual era a R por ser uma separação inédita e não haver informações suficientes na literatura para comparar”, explica Bruno Amaral. “Cruzamos dados teóricos e experimentais para identificar os enantiômeros e a colaboração do professor João Batista, da Unifesp, foi fundamental”.
“As colaborações com diferentes universidades e laboratórios são fundamentais para conseguirmos avançar com a ciência de forma complementar”, comenta Katlin Massirer, pesquisadora do Centro de Biologia Molecular e Engenharia Genética (CBMEG-Unicamp) e coordenadora do estudo.
Apesar da boa permeabilidade e solubilidade adequada, a molécula ML-SA1(S) apresenta baixa estabilidade para ser usada como um medicamento para induzir a autofagia. No entanto, os novos resultados apresentados sobre ela podem fornecer informações relevantes para o desenvolvimento de medicamentos estáveis, que sirvam para induzir o processo de autofagia em seres humanos. Fármacos para essa finalidade apresentam grande potencial terapêutico e econômico.
Enantiômeros são moléculas com a mesma estrutura e o mesmo número de átomos, porém, desviam o plano da luz polarizada para lados diferentes. Essa diferença é fundamental do ponto de vista químico e tem efeitos relevantes no funcionamento celular. Seus efeitos já são explorados pela indústria farmacêutica e alimentícia.
Um trágico exemplo envolvendo enantiômeros foi o caso do medicamento talidomida, que demonstra a importância de se avaliar cuidadosamente as diferenças funcionais e toxicológicas dos enantiômeros presentes nos compostos antes de serem comercializados como medicamentos.
A pesquisa recebeu financiamento da FAPESP por meio de três projetos e também teve apoio de outras instituições. O artigo pode ser lido em: [link do artigo].
Informações da Agência FAPESP